Antibioticaresistentie is wereldwijd een groeiende bedreiging voor de volksgezondheid. De Wereldgezondheidsorganisatie waarschuwde onlangs dat hierdoor eenvoudige bacteriële infecties zoals longontsteking, moeilijker te behandelen worden. Wanneer bacteriën herhaaldelijk zijn blootgesteld aan antibiotica passen ze zich aan, waardoor medicijnen die voorheen effectief waren nu nutteloos zijn. Het vaststellen van resistentie kan nu dagen in beslag nemen en vereist vaak dure laboratoriumapparatuur. Het iGEM-team van de Rijksuniversiteit Groningen heeft hiervoor een oplossing gevonden: de CRIKIT.
(iGEM, international Genetically Engineered Machine, is een internationale, jaarlijkse wedstrijd waarin multidisciplinaire teams van studenten met behulp van de nieuwste Synthetische Biologie technieken een biologische machine bouwen.)
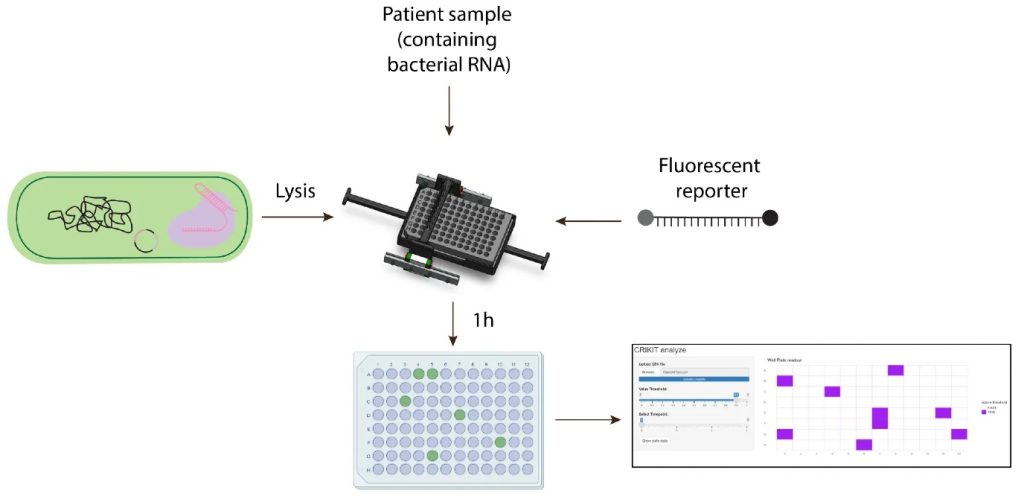
De CRIKIT werkt met CRISPR Cas13-technologie, die zich richt op het RNA van antibioticaresistente bacteriën. Teamlid Mihai Frăcea legt uit: “Je kunt Cas13 zien als een schaar met een GPS. Het Cas13-enzym is de schaar; het knipt en geeft ons ook een fluorescerend signaal.” Dit lichtsignaal geeft aan wanneer antibioticaresistentie wordt gedetecteerd. Maar de schaar heeft aanwijzingen nodig om te weten waar hij moet knippen. Daarvoor is een ‘guide-RNA’ (gRNA), ook wel ‘spacer’ genoemd, nodig. “De spacer is de GPS. Hij vertelt de schaar waar die heen moet.” Door andere soorten spacers te gebruiken, kan de CRIKIT verschillende resistentiegenen detecteren.
De plaatlezer is klein van ontwerp, maar zit boordevol snufjes. Hij maakt gebruik van enkele eenvoudige ledlampjes, fotosensoren en filters om de fluorescentie te meten. Daarnaast is het kastje uitgerust met bluetooth en wifi om de meetresultaten naar een computer te sturen. Daar analyseert de eigen software van het team de gegevens en geeft een overzicht van de genen van de bacteriële infectie van de patient die resistent zijn tegen antibiotica.
Naast tijdwinst streeft het team ernaar om hun ontwerp voor iedereen beschikbaar te maken. “Het detecteren van fluorescentie is in de meeste gevallen erg duur”, legt Frăcea uit. Hij schat dat plaatlezers tot wel een miljoen euro kunnen kosten. “Het is een enorme doos en hij is erg zwaar. Je kunt hem dus niet echt meenemen en verplaatsen, en hij is niet echt inzetbaar in het veld.” Om dat probleem op te lossen, besloot het team om hun eigen versie van een plaatlezer te bouwen. Met trots schrijft Frăcea het ontwerp van de hardware van CRIKIT toe aan teamlid Dilara Yigit. “Het is gewoon een doos waarin je je monsters stopt, en hij is 3D-geprint, dus vrij goedkoop”, zegt Frăcea. “Het is in feite een plaatlezer die in plaats van een miljoen maar een paar honderd euro kost. En je kunt hem zelfs hergebruiken.”
Hoewel het team zich momenteel richt op antibioticaresistentie, kan het basisontwerp van CRIKIT (het detecteren van genetisch materiaal) ook voor andere doeleinden buiten de gezondheidszorg worden gebruikt. “Onze kit kan heel gemakkelijk worden aangepast voor bijvoorbeeld de landbouw”, stelt Frăcea. “We kunnen bijvoorbeeld gemakkelijk infecties of bacteriën detecteren die gewassen aantasten.”
Lees het hele bericht op de site van de RUG.